Klinische Kooperationseinheit Neuroonkologie (Prof. Dr. Wolfgang Wick)
Forschungsschwerpunkte
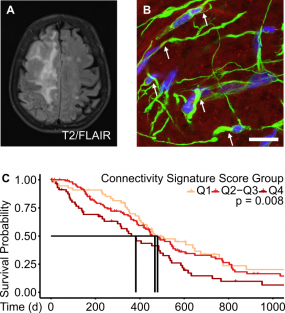
(A) T2-FLAIR-MRT eines Glioblastom Patienten. (B) Heterogenität im Glioblastom. Veranschaulicht anhand unterschiedlicher Längen der Tumormikrotubes (grün, Nestin) einzelner Zellen (blau, DAPI). (C) Die Konnektivitätssignatur ist ein prognostischer Glioblastommarker
© dkfz.de
Die Forschung der klinischen Kooperationseinheit (KKE) Neuroonkologie hat das übergeordnete Ziel, die biologischen Mechanismen, die dem Glioblastom und dem primären ZNS-Lymphom, sowie deren Therapieresistenz zugrunde liegen, umfassender zu verstehen. Ein weiterer Schwerpunkt liegt auf der Identifizierung und Validierung von diagnostischen, prognostischen und prädiktiven Biomarkern für Leitlinien und studienbasiert-zielgerichtete Therapien. Die aus dieser Forschung hervorgehenden potenziellen Angriffspunkte für eine gezielte therapeutische Intervention werden präklinisch untersucht und ultimativ in nachfolgenden klinischen Studien getestet. Die aktuelle und zukünftige Ausrichtung der Forschung in der KKE Neuroonkologie folgt einem translationalem "bench-to-bedside" Ansatz, welcher sich an aus dem klinischen Neuroonkologie-Programm abgeleiteten zentralen Forschungsfragen orientiert, und hat das Ziel, die vorklinischen Erkenntnisse rasch in die Klinik zu übersetzen.
Die KKE Neuroonkologie hat sechs Forschungsschwerpunkte:
- Molekulare Dechiffrierung von Glioblastom-Netzwerken und insbesondere der Rolle des Tumornetworkgenes CHI3L1. Mit der bahnbrechenden Entdeckung multizellulärer Glioblastom-Netzwerke ergab sich gleichzeitig ein neuartiger Ansatz um das Wachstum und die Resistenz von Hirntumoren zu erklären. Mit dem Ziel, die molekularen Grundlagen dieser Netzwerke zu entschlüsseln, haben wir eine transkriptomische Signatur für verbundene Glioblastomzellen durch Einzelzell- und Bulk-RNA-Sequenzierung von In-vitro- und In-vivo-Modellen sowie Patientengewebe aus klinischen Studien etabliert. Chitinase-3-like 1 (CHI3L1), einen molekularen Marker mit funktioneller Relevanz für Tumorzellnetzwerke und potenzieller therapeutischer Anwendbarkeit, beim Glioblastom identifiziert und seine Bedeutung validiert. Diese Entdeckungen haben den Weg für weitere Folgeprojekte zum genauen Wirkmechanismus von CHI3L1 und zur Relevanz anderer mutmaßlicher Tumornetzwerk-relevanter Gene geebnet
- Identifizierung von Biomarkern und eingehenderes Verständnis von Resistenzmechanismen gegen zielgerichtete Wirkstoffe beim Glioblastom. Dieser Forschungszweig zielt darauf ab, die molekularen Mechanismen mehrerer zielgerichteter Therapien wie z.B. Temsirolimus, einem mTOR-Inhibitor, und Atezolizumab, einem Anti-PD-L1-Antikörper, besser zu verstehen. Darüber hinaus erforschen wir zelluläre Signaltransduktionswege, die an der erworbenen Resistenz beteiligt sind, wie z.B. der TP53-Signalweg im Kontext einer Chemotherapie. Es besteht ein hohes Interesse daran, die Wechselwirkung zwischen Bestrahlung und den molekular zielgerichteten Behandlungen zu verstehen, welche im Rahmen der NCT Neuro Master Match (N2M2) Umbrella-Studie und weiterer, von der klinischen Sektion der Neuroonkologie koordinieren, randomisierten Studien verabreicht werden. Hierzu wird das Patientengewebe mit zahlreichen Omics-Technologien zunächst analysiert und hervorgehende Marker validiert.
- Diagnosestellung von Hirnerkrankungen mittels Flüssigbiopsien. Das Flüssigbiopsie-Programm für maligne Hirntumoren umfasst einen integrativen Multiomik-Ansatz in dem Patientenliquor mittels Zytologie, Proteomik und Sequenzierung von zirkulierender Tumor-DNA analysiert wird. Die Proteomik ermöglicht die Identifizierung von Liquor-Biomarkern, die für Glioblastome im Vergleich zu anderen malignen Gehirnerkrankungen spezifisch sind, sowie die Gruppierung von Glioblastom-Patienten in prognostisch unterschiedliche Cluster. Die ctDNA-Sequenzierungsplattform, die ein Panel von 170 Hirntumor-relevanten Genen umfasst, erlaubt Glioblastom-spefzifische Mutations- und Kopienzahlprofile zu erkennen, wodurch auf Gewebebiopsien zur Diagnose und Tumorentwicklung verzichtet werden kann.
- Molekulare Fingerabdrücke und phänotypische Merkmale von Glioblastom-Stammzellen.
- Entwicklung von Immuntherapien für Hirntumore. Antigene die vor und nach einer Glioblastom-Therapie exklusiv oder deutlich hochreguliert sind im Vergleich zu gesunden Geweben, werden sowohl mit Immun-Checkpoint-Inhibitoren, Tumorimpfstoffen als auch CAR-T-Zell adressiert. Wir untersuchen insbesondere Interventionstherapien für PD-L1, IDH1 R132H sowie weitere, nach einer Leitlinientherapie hochreguliere Targets.
- Charakterisierung von primären Lymphomen des Zentralnervensystems (PCNSL). In bereits durchgeführten Studien haben wir mittels diverser genetischer Analysen (unter anderem Whole Exome/Genome Sequencing, 850k-Analyse) von PCNSL-Proben molekulare Signaturen der ZNS-Lymphomgenese entschlüsselt. Jedoch ist die funktionelle Bedeutung von vielen der neu entdeckten genetischen Veränderungen noch unzureichend verstanden. Die Befunde werden daher in Lymphom-Zelllinien in vitro und in vivo unter Verwendung von Xenograft-Modellen modelliert und die phänotypischen und molekularen Folgen dieser genetischen Veränderungen untersucht. In vitro-Medikamentenscreenings und CAR-T-Zell-Engineering ermöglichen weiterhin die Applikabilität neuartiger zielgerichteter Therapien gegen diese Veränderungen. Nach einer erfolgreichen Evaluierung im präklinischen Mausmodell sollen die Erkenntnisse möglichst früh in klinische Studien überführt werden.
Unsere KKE Neuroonkologie arbeitet eng mit der KKE Neuroimmunologie und Hirntumorimmunologie (D170) und KKE Neuropathologie (B300) zusammen. Zudem existieren vitale Kooperationen mit der Abteilung für Hämatologie, Onkologie und Rheumatologie sowie mit den Abteilungen für experimentelle Bildgebung an der Universität Heidelbeg und dem DKFZ. Darüber hinaus bestehen enge Kooperationen innerhalb des UNITE-Glioblastom-Konsortiums (https://www.unite-glioblastoma.de/). Die Einbindung in ein nationales sowie internationales Netzwerk unterstützt die Forschung zusätzlich und ist für den schnellen Transfer von Forschungsergebnissen in die Klinik von größter Bedeutung.
Ausgewählte Publikationen
- Hausmann, D.; Hoffmann, D.C.; Venkataramani, V.; Jung, E.; Horschitz, S.; Tetzlaff, S.K.; Jabali, A.; Hai, L.; Kessler, T.; Azoŕin, D.D.; Weil, S.; Kourtesakis, A.; Sievers, P.; Habel, A.; Breckwoldt, M.O.; Karreman, M.A.; Ratliff, M.; Messmer, J.M.; Yang, Y.; Reyhan, E.; Wendler, S.; Löb, C.; Mayer, C.; Figarella, K.; Osswald, M.; Solecki, G.; Sahm, F.; Garaschuk, O.; Kuner, T.; Koch, P.; Schlesner, M; Wick, W.; Winkler, F. (2023): Autonomous rhythmic activity in glioma networks drives brain tumour growth. In Nature 613 (7942), pp. 179–186. DOI: 10.1038/s41586-022-05520-4.
- Hai, L.; Hoffmann, D.C.; Mandelbaum, H.; Xie, R.; Ito, J.; Jung, E. et al. (2021): A connectivity signature for glioblastoma.
- Kaulen, L.D.; Erson-Omay, E.Z.; Henegariu, O.; Karschnia, P.; Huttner, A.; Günel, M.; Baehring, J.M. (2021): Exome sequencing identifies SLIT2 variants in primary CNS lymphoma. In British journal of haematology 193 (2), pp. 375–379. DOI: 10.1111/bjh.17319.
- Schmid, D.; Warnken, U.; Latzer, P.; Hoffmann, D.C.; Roth, J.; Kutschmann, S.; Jaschonek, H.; Rübmann, P.; Foltyn, M.; Vollmuth, P.; Winkler, F.; Seliger, C.; Felix, M.; Sahm, F.; Haas, J.; Reuss, D.; Bendszus, M.; Wildemann, B.; Deimling, A.v.; Wick, W.; Kessler, T. (2021): Diagnostic biomarkers from proteomic characterization of cerebrospinal fluid in patients with brain malignancies. In Journal of neurochemistry 158 (2), pp. 522–538. DOI: 10.1111/jnc.15350.
- Platten, M.; Bunse, L.; Wick, A.; Bunse, T.; Le Cornet, L.; Harting, I.; Sahm, F.; Sanghvi, K.; Tan, C.L.; Poschke, I.; Green, E.; Justesen, S.; Behrens, G.A.; Breckwoldt, M.O.; Freitag, A.; Rother, L.M.; Schmitt, A.; Schnell, O.; Hense, J.; Misch, M.; Krex, D.; Stevanovic, S.; Tabatabai, G.; Steinbach, J.P.; Bendszus, M.; Deimling, A.v.; Schmitt, M.; Wick, W. (2021): A vaccine targeting mutant IDH1 in newly diagnosed glioma. In Nature 592 (7854), pp. 463–468. DOI: 10.1038/s41586-021-03363-z.
- Wick, W.; Dettmer, S.; Berberich, A.; Kessler, T.; Karapanagiotou-Schenkel, I.; Wick, A.; Winkler, F.; Pfaff, E.; Brors, B.; Debus, J.; Unterberg, A.; Bendszus, M.; Herold-Mende, C.; Eisenmenger, A.; Deimling, A.v.; Jones, D.T.W.; Pfister, S.M.; Sahm, F.; Platten, M. (2019): N2M2 (NOA-20) phase I/II trial of molecularly matched targeted therapies plus radiotherapy in patients with newly diagnosed non-MGMT hypermethylated glioblastoma. In Neuro-oncology 21 (1), pp. 95–105. DOI: 10.1093/neuonc/noy161.
- Berberich, A.; Kessler, T.; Thomé, C.M.; Pusch, S.; Hielscher, T.; Sahm, F.; Oezen, I.; Schmitt, L.M.; Ciprut, S.; Hucke, N. Ruebmann, P.; Fischer, M.; Lemke, D.; Breckwoldt, M.O.; Deimling, A.v.; Bendszus, M.; Platten, M.; Wick, W. (2019): Targeting Resistance against the MDM2 Inhibitor RG7388 in Glioblastoma Cells by the MEK Inhibitor Trametinib. In Clinical cancer research : an official journal of the American Association for Cancer Research 25 (1), pp. 253–265. DOI: 10.1158/1078-0432.CCR-18-1580.
- Kessler, T. Sahm, F.; Sadik, A.; Stichel, D.; Hertenstein, A.; Reifenberger, G.; Zacher, A.; Sabel, M.; Tabatabai, G.; Steinbach, J.; Sure, U.; Krex, D.; Grosu, A.L.; Bewerunge-Hudler, M.; Jones, D.; Pfister, S.M.; Weller, M.; Opitz, C.; Bendszus, M.; Deimling, A.v.; Platten, M.; Wick, W. (2018): Molecular differences in IDH wildtype glioblastoma according to MGMT promoter methylation. In Neuro-oncology 20 (3), pp. 367–379. DOI: 10.1093/neuonc/nox160.
